新しいハイパラメータサイトメトリー装置を使用できるようになった方々、15 以上のパラメータパネルがあなたを呼んでいます。こんなにたくさんのマーカーをどう扱いますか? どの表現型を見ればよいかも分からないのに、疾患モデルにおけるコントロールと処理済みサンプルとの差をどうやって見つけていけばよいのでしょう?
1 つの方法は次元縮退アルゴリズムを使用することです。このアルゴリズムは、データ構造を保ちながら、N 次元のデータ空間を 2 次元に縮退します。
FlowJo V10 には、tSNE (t-Distributed Stochastic Neighbor Embedding) という次元縮退アルゴリズムのプラグインが搭載されています。tSNE アルゴリズムは、ユーザー定義のサイトメトリーパラメータ群から 2 つのパラメータを新たに算出します。tSNE で作成されたパラメータは、高次元の生データにおいて接近していたデータポイントどうしが縮退データ空間でも接近するように最適化されています。
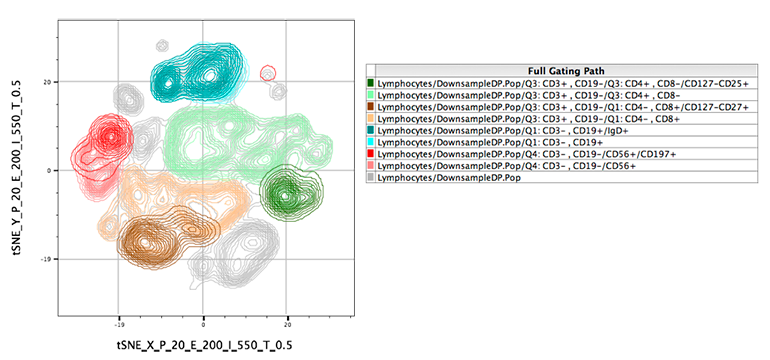
図 1. tSNE による 2 次元データ空間への縮退後の 15 色フローサイトメトリーパネルの例。手動ゲーティングにより得られた既知の表現型の集団を、FlowJo レイアウトエディタで tSNE 空間に重ね合わせています。異なる表現型を持つイベントサブセットがそれぞれクラスタ化し、大陸のような構造で異なる領域に集中している様子が分かります。
tSNE は効果的な表示法ですが、このアルゴリズムは計算に頼る部分が多く、また出力も決定的なものではありません。つまり、1) 適正な時間内に計算が終わるようにするには、アルゴリズムに入力するイベント数を制限する必要があります。また、2) 2 つの別々のサンプルでアルゴリズムを実行する場合など、アルゴリズムを複数回実行すると、tSNE で作成される 2 次元データ空間がサンプル間で異なってきます。
したがって、サンプルを効果的に比較する唯一の方法は、イベント数を減らし (ダウンサンプリング処理)、サンプルを 1 つの FCS ファイルにまとめてから (連結処理)、連結データセットで次元縮退を行うことです。
連結ステップでは、キーワード/値のペアに基づいて、任意で新しい導出パラメータを作成できます。こうしておくと、tSNE で作成された共通の次元縮退データ空間内で、異なるサンプルや異なる実験条件 (タイムポイント、処理グループ、シミュレーションなど) からイベントを分離できます。
ワークフローの例 :
シグナル応答とサイトカイン発現を誘発する薬剤で処理する時間を変えた、4 つのサンプルがある場合の解析
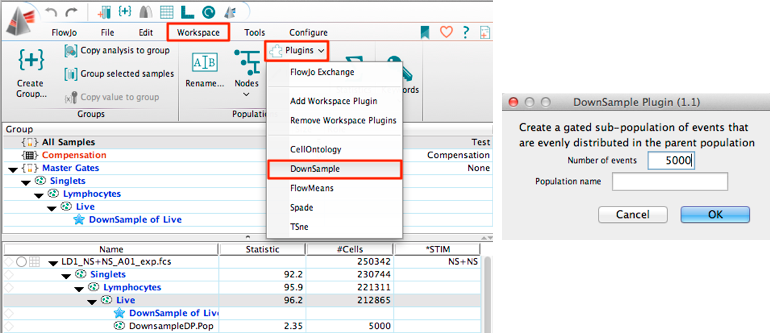
図 2 DownSample プラグインの起動
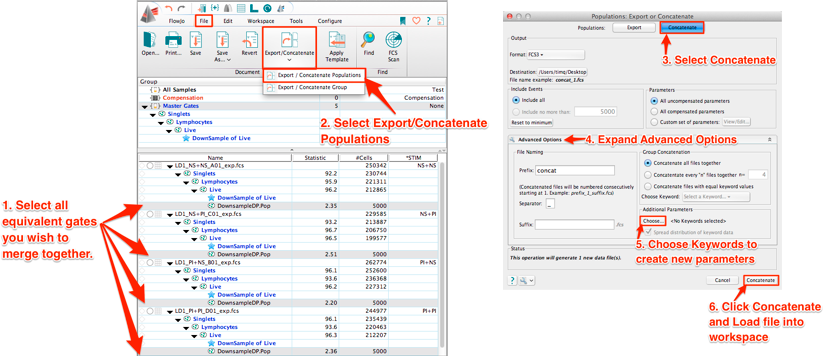
図 3.集団の連結
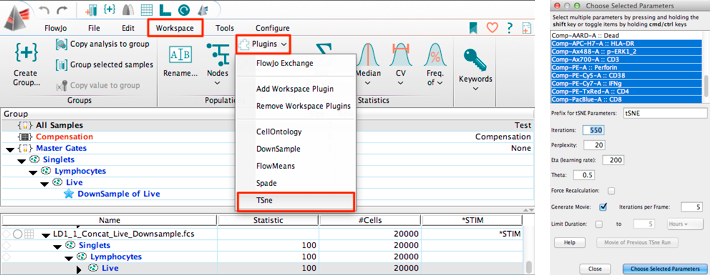
図 4.tSNE プラグインの起動
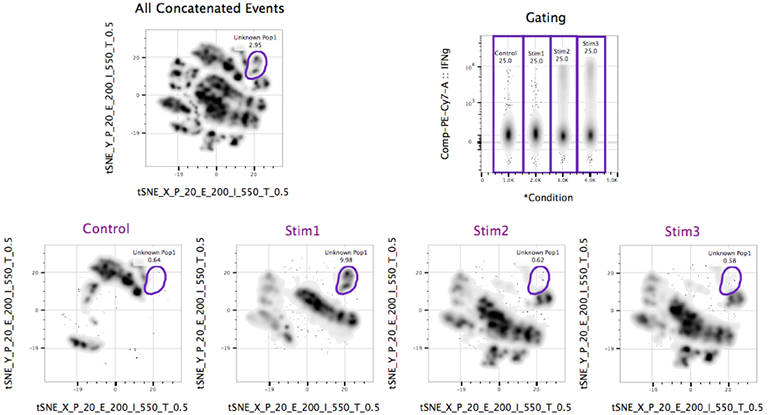
図 5.キーワードベースの導出パラメータによるゲーティングで連結ファイル内の個々のサンプルを分離
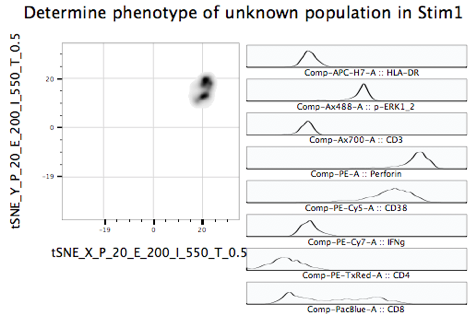
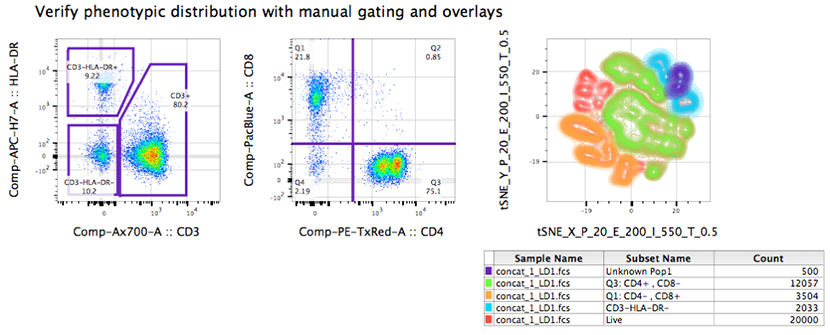
図 6.マルチグラフヒストグラムプロットによる表現型発現の判定と手動作成ゲートによる検証
BD, the BD Logo and all other trademarks are trademarks of Becton, Dickinson and Company or its affiliates. © BD. All rights reserved.